
Nos recherches portent sur l’étude de la bactérie phytopathogène Xanthomonas campestris pv. campestris (Xcc), agent causal de la pourriture noire chez les Brassicaceae. Ce pathogène infecte des cultures importantes sur le plan agricole, telles que le chou, le brocoli, le colza et le chou-fleur, ainsi que la plante modèle Arabidopsis thaliana.
Nos travaux se concentrent sur trois processus majeurs régissant les interactions entre les plantes et les pathogènes :
● Les réponses immunitaires des plantes à Xanthomonas, en particulier dans les hydathodes et le système vasculaire.
● Les bases génétiques de la pathogénie de Xanthomonas et son adaptation à divers tissus végétaux et conditions environnementales.
● Le déterminisme génétique de la gamme d'hôtes induit par l'effectome de type III de Xanthomonas.
Nous utilisons un large éventail d'approches, notamment de la génétique moléculaire, de la biologie synthétique, des technologies « omiques » et des criblages génétiques à haut débit pour atteindre ces objectifs.
Contact: Laurent NOEL
THÈMES DE RECHERCHE
Réponses immunitaires tissu végétal dépendantes face à Xanthomonas
Chercheurs principaux : Laurent NOEL et Jean-Marc ROUTABOUL
Nous étudions les réponses moléculaires, développementales et physiologiques des plantes qui leur confèrent une résistance ou une sensibilité à l'infection par Xanthomonas.
Nos travaux ont permis une analyse approfondie de l'anatomie, de la physiologie et de l'immunité des hydathodes, soulignant leur identité distincte par rapport aux limbes foliaires, cela grâce à des approches de métabolomiques, de transcriptomiques et de génomiques fonctionnelles. Les résultats ont montré que les hydathodes accumulent de l’auxines, captent des nutriments et présentent des fonctions physiologiques uniques. De plus, des travaux sur les hydathodes du chou-fleur ont révélé que les effecteurs de type III de Xcc suppriment fortement les réponses immunitaires dans ce tissu.
A) Des échantillons de dents foliaires contenant des hydathodes et de limbes foliaires ont été prélevés par macro dissection sur des feuilles de plantes âgées de 9 à 10 semaines, puis soumis à un séquençage d'ARN. (B) Catégorisation fonctionnelle des 1 086 gènes différentiellement exprimés avec une annotation fonctionnelle (461/625 induits/réprimés dans les hydathodes). Les gènes différentiellement exprimés ont été principalement annotés à l'aide de la classification d'analyse MapMan disponible sur le site BAR. L'échelle de couleurs indique la moyenne log2 Fold Change (FC) des gènes appartenant à la catégorie fonctionnelle donnée qui étaient globalement soit plus exprimés dans les hydathodes (en rouge), soit dans le limbe foliaire (en bleu). (C,D) Échantillons frais préparés par cryogénie observés au microscope électronique à balayage. (E) Observation en Nomarsky d'un échantillon d'hydathode clarifié. Notez les nombreux vaisseaux du xylème.
Nous étudions la réponse d'Arabidopsis conférant une résistance ou une sensibilité à l'infection par Xanthomonas en exploitant des criblages génétiques directs réalisés à partir de la collection de mutants HEM.
(a) Photo de 104 lignées Hem sur les 900 disponibles. (b) Représentation sous forme de diagramme en violon de la distribution du nombre de mutations par lignée.
Nous nous intéressons à l'immunité établie dans les hydathodes et le système vasculaire des plantes, lesquelles impactent l'entrée et la propagation des agents pathogènes respectivement. Nous étudions en particulier le rôle de la disponibilité en eau dans l'établissement de la résistance et de la sensibilité à Xanthomonas. Cet axe est soutenu par deux subventions de l'ANR (VIP et HIRAQUIM).
Adaptation de Xanthomonas à son environnement
Chercheuse principale : Alice BOULANGER
L'adaptation des agents pathogènes aux changements environnementaux est essentielle à leur virulence. Les analyses transcriptomiques ont permis de mieux comprendre le stress et l'état nutritionnel de Xanthomonas aux premiers stades de l'infection et ont mis en évidence diverses stratégies de virulence dépendantes de HrpG chez les souches de Xanthomonas.
Représentation schématique des principales réponses transcriptionnelles de Xcc au cours de la phase précoce de colonisation de l'hydathode : les gènes correspondant aux objets bleus et rouges sont respectivement réprimés et induits entre 4 et 72 heures après l'inoculation. Les gènes correspondant aux objets gris ne sont pas exprimés de manière différentielle. T3SS : système de sécrétion de type III ; T3E : effecteur de type III ; T2SS : système de sécrétion de type II ; LPS : lipopolysaccharide ; PCWDE : enzymes dégradant la paroi cellulaire végétale ; T4P : pilus de type IV. Figure dessinée à l'aide de biorender.
Nous effectuons des criblages RB-TnSeq à haut débit afin d'identifier les gènes de Xcc impliqués dans la colonisation des tissus végétaux et l'adaptation à divers environnements, en mettant particulièrement l'accent sur les protéines hypothétiques (HP) et les régulateurs transcriptionnels (TR) non caractérisés.
Schéma expérimental
Mécanismes de virulence dépendants du type III et déterminisme génétique de la gamme d'hôtes
Chercheurs principaux : Corinne AUDRAN et Carlos ZARATE CHAVES
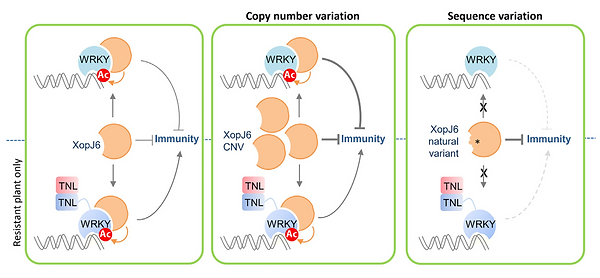
Notre équipe étudie les rôles individuels et collectifs des effecteurs de type III (T3E) de Xanthomonas. Nous avons identifié XopJ6, un homologue de l’effecteur PopP2 de Ralstonia pseudosolanacearum, comme un facteur clé déclenchant la résistance aux maladies chez le chou-fleur et Arabidopsis. Notre étude a révélé comment XopJ6 peut soit déclencher, soit contourner l'immunité des plantes, et comment l'augmentation du nombre de copies de xopJ6 favorise la virulence.
En parallèle, nous nous sommes lancés dans la déconstruction de l'ensemble de l'effectome de Xcc, ce qui a permis d'obtenir une souche non pathogène dépourvue d'effecteurs. Il est intéressant de noter que sa pathogénie peut être restaurée sur Arabidopsis grâce à un effectome synthétique composé de seulement six effecteurs. L'équipe met actuellement en œuvre un pipeline de biologie synthétique combiné à des mesures de fitness à haut débit afin d'évaluer la fonctionnalité du mini-effectome sur les plantes hôtes et non hôtes.
Les TALE (Transcription Activator-Like Effectors) sont des protéines T3E spécifiques à Xanthomonas qui modulent l'expression des gènes végétaux afin de faciliter l'infection. Leur rôle crucial dans la pathogénie de Xanthomonas a été démontré à l'aide de la technologie d'interférence CRISPR, qui a permis une répression précise de l'expression de gènes tal dans différentes souches bactériennes.
Chez Xcc, la contribution spécifique des TALE est actuellement étudiée à la fois dans des plantes hôtes naturelles et dans des espèces non hôtes, dans le but d'identifier leurs cibles végétales directes et de clarifier leur double rôle en tant que facteurs de virulence ou d'avirulence.
En comprenant les mécanismes de virulence et la gamme d'hôtes de Xanthomonas, nous visons à prédire comment ces agents pathogènes peuvent émerger dans de nouveaux environnements et s'adapter à de nouvelles plantes hôtes. Ces travaux représentent une base pour le développement de modèles prédictifs permettant d'anticiper les épidémies d'agents pathogènes et d'atténuer l'impact des maladies bactériennes dans l'agriculture.
Collaborations
-
Adam Bogdanove, Cornell University, NY
-
Jian-Min Zhou, Beijing, China
-
Jennifer Lewis, UC Berkeley, CA.
-
Anne Genissel, INRA Versailles, France
-
Richard Berthomé, Laurent Deslandes and Fabrice Roux, LIPM Toulouse, France
-
Boris Szurek & Ralf Koebnik, IRD Montpellier, France
-
Matthieu Barret, Nicolas Chen and Marie-Agnès Jacques, INRA Angers, France
-
Lionel Gagnevin and Olivier Pruvost, CIRAD La Réunion, France
Financements en cours
-
ANR VIP (2024-2029) ANR-24-CE20-5527-01 Immunité vasculaire des plantes/plant vascular immunity. L. Noël (Coordinator, LIPME Toulouse), L. Navarro (IBENS Paris), N. Peeters (LIPME Toulouse), Marie-Laure Martin Magnette (IPS2 Saclay). 818k€ (360k€ for LIPME).
-
ANR HIRAQUIM (2023-2028) ANR-23-CE20-0022-02 Rôle régulateur des protéines HIR localisées dans les nanodomaines membranaires dans l'immunité dépendante des aquaporines chez les plantes/Regulatory role of the membrane nanodomain‐localized proteins HIR in the aquaporin‐dependent plant immunity. Enric Zelazny (Coodinator, IPSIM Montpellier), L. Noël (LIPME Toulouse). 547k€ (214k€ for LIPME).
-
Financement sur projet Département SPE INRAE : XccOnFire (2025-2026), A. Boulanger, 30 k€
-
Research networks INRAE SPE Department: FNX = French Network on Xanthomonads. M-A Jacques, R. Koebnik, O. Pruvost and L. Noël, coordinators, 5 k€/year.




PUBLICATIONS
2025
Routaboul, J.-M., Corso, M., & Lepiniec, L. (2025). Flavonoid characterization : It takes more than Arabidopsis seed colours! Planta, 262(5), 112.
https://doi.org/10.1007/s00425-025-04828-5
2024
Routaboul, J.-M., Bellenot, C., Olympio, A., Clément, G., Citerne, S., Remblière, C., Charvin, M., Franke, L., Chiarenza, S., Vasselon, D., Jardinaud, M.-F., Carrère, S., Nussaume, L., Laufs, P., Leonhardt, N., Navarro, L., Schattat, M., & Noël, L. D. (2024). Arabidopsis hydathodes are sites of auxin accumulation and nutrient scavenging. The Plant Journal: For Cell and Molecular Biology. https://doi.org/10.1111/tpj.17014
Quiroz Monnens, T., Roux, B., Cunnac, S., Charbit, E., Carrère, S., Lauber, E., Jardinaud, M.-F., Darrasse, A., Arlat, M., Szurek, B., Pruvost, O., Jacques, M.-A., Gagnevin, L., Koebnik, R., Noël, L. D., & Boulanger, A. (2024). Comparative transcriptomics reveals a highly polymorphic Xanthomonas HrpG virulence regulon. BMC Genomics, 25(1), 777.
https://doi.org/10.1186/s12864-024-10684-6
Carrère, S., Routaboul, J.-M., Savourat, P., Bellenot, C., López, H., Sahoo, A., Quiroz Monnens, T., Ricou, A., Camilleri, C., Declerck, N., Laufs, P., Mercier, R., & Noël, L. D. (s. d.). A fully sequenced collection of homozygous EMS mutants for forward and reverse genetic screens in Arabidopsis thaliana. The Plant Journal, 2024 Jul 29. https://doi.org/10.1111/tpj.16954
Koebnik, R., Cesbron, S., Chen, N. W. G., Fischer-Le Saux, M., Hutin, M., Jacques, M.-A., Noël, L. D., Perez-Quintero, A., Portier, P., Pruvost, O., Rieux, A., & Szurek, B. (2024). Celebrating the 20th Anniversary of the First Xanthomonas Genome Sequences – How Genomics Revolutionized Taxonomy, Provided Insight into the Emergence of Pathogenic Bacteria, Enabled New Fundamental Discoveries and Helped Developing Novel Control Measures – A Perspective from the French Network on Xanthomonads. Zenodo. https://doi.org/10.5281/zenodo.10683038
Lauber, E., González-Fuente, M., Escouboué, M., Vicédo, C., Luneau, J. S., Pouzet, C., Jauneau, A., Gris, C., Zhang, Z.-M., Pichereaux, C., Carrère, S., Deslandes, L., & Noël, L. D. (2024). Bacterial host adaptation through sequence and structural variations of a single type III effector gene. iScience, 27(3), Article 3. https://doi.org/10.1016/j.isci.2024.109224
Monnens, T. Q., & Boulanger, A. (2024). A large scale bacterial attraction assay : A new quantitative bacterial migration assay suitable for genetic screens. PLOS ONE, 19(6), e0305037. https://doi.org/10.1371/journal.pone.0305037
2023
Marzorati, F., Rossi, R., Bernardo, L., Mauri, P., Silvestre, D. D., Lauber, E., Noël, L. D., Murgia, I., & Morandini, P. (2023). Arabidopsis thaliana Early Foliar Proteome Response to Root Exposure to the Rhizobacterium Pseudomonas simiae WCS417. Molecular Plant-Microbe Interactions: MPMI, 36(11), 737‑748. https://doi.org/10.1094/MPMI-05-23-0071-R
Talbi, N., Fokkens, L., Audran, C., Petit-Houdenot, Y., Pouzet, C., Blaise, F., Gay, E. J., Rouxel, T., Balesdent, M.-H., Rep, M., & Fudal, I. (2023). The neighbouring genes AvrLm10A and AvrLm10B are part of a large multigene family of cooperating effector genes conserved in Dothideomycetes and Sordariomycetes. Molecular Plant Pathology. https://doi.org/10.1111/mpp.13338
Zárate-Chaves, C. A., Audran, C., Medina Culma, C. A., Escalon, A., Javegny, S., Gagnevin, L., Thomas, E., Pimparé, L.-L., López, C. E., Jacobs, J. M., Noël, L. D., Koebnik, R., Bernal, A. J., & Szurek, B. (2023). CRISPRi in Xanthomonas demonstrates functional convergence of transcription activator-like effectors in two divergent pathogens. The New Phytologist, 238(4), 1593‑1604. https://doi.org/10.1111/nph.18808
2022
You, Y., Koczyk, G., Nuc, M., Morbitzer, R., Holmes, D. R., von Roepenack-Lahaye, E., Hou, S., Giudicatti, A., Gris, C., Manavella, P. A., Noël, L. D., Krajewski, P., & Lahaye, T. (2022). The eINTACT system dissects bacterial exploitation of plant osmosignalling to enhance virulence. Nature Plants, 1‑14. https://doi.org/10.1038/s41477-022-01302-y
Luneau, J. S., Baudin, M., Quiroz Monnens, T., Carrère, S., Bouchez, O., Jardinaud, M.-F., Gris, C., François, J., Ray, J., Torralba, B., Arlat, M., Lewis, J. D., Lauber, E., Deutschbauer, A. M., Noël, L. D., & Boulanger, A. (2022). Genome-wide identification of fitness determinants in the Xanthomonas campestris bacterial pathogen during early stages of plant infection. The New Phytologist. https://doi.org/10.1111/nph.18313
Bellenot, Caroline, Jean-Marc Routaboul, Patrick Laufs, et Laurent D. Noël. « Hydathodes ». Current Biology 32, no 14 (25 juillet 2022): R763‑64. https://doi.org/10.1016/j.cub.2022.06.014.
Bellenot, C., Carrère, S., Gris, C., Noël, L. D., & Arlat, M. (2022). Genome Sequences of 17 Strains from Eight Races of Xanthomonas campestris pv. Campestris. Microbiology Resource Announcements, 11(7), e0027922. https://doi.org/10.1128/mra.00279-22
Luneau, J. S., Noël, L. D., Lauber, E., & Boulanger, A. (2022). A β-glucuronidase (GUS) Based Bacterial Competition Assay to Assess Fine Differencesin Fitness during Plant Infection. Bio-protocol, 12(13), e3776. https://doi.org/10.21769/BioProtoc.3776
Dubrow, Zoe, Sara Carpenter, Morgan E. Carter, Ayress Grinage, Carine Gris, Emmanuelle Lauber, Jules Butchacas, et al. « Cruciferous Weed Isolates of Xanthomonas Campestris Yield Insight into Pathovar Genomic Relationships and Genetic Determinants of Host- and Tissue-Specificity ». Molecular Plant-Microbe Interactions: MPMI, 10 mai 2022. https://doi.org/10.1094/MPMI-01-22-0024-R.
Luneau, Julien S., Aude Cerutti, Brice Roux, Sébastien Carrère, Marie-Françoise Jardinaud, Antoine Gaillac, Carine Gris, et al. « Xanthomonas Transcriptome inside Cauliflower Hydathodes Reveals Bacterial Virulence Strategies and Physiological Adaptations at Early Infection Stages ». Molecular Plant Pathology 23, no 2 (février 2022): 159‑74. https://doi.org/10.1111/mpp.13117.
2021
Zhu, Xiaoyang, Julie Mazard, Eugénie Robe, Sarah Pignoly, Marielle Aguilar, Hélène San Clemente, Emmanuelle Lauber, Richard Berthomé, et Jean-Philippe Galaud. « The Same against Many: AtCML8, a Ca2+ Sensor Acting as a Positive Regulator of Defense Responses against Several Plant Pathogens ». International Journal of Molecular Sciences 22, no 19 (28 septembre 2021): 10469. https://doi.org/10.3390/ijms221910469.
2020
Jauneau, Alain, Aude Cerutti, Marie-Christine Auriac, et Laurent D. Noël. « Anatomy of Leaf Apical Hydathodes in Four Monocotyledon Plants of Economic and Academic Relevance ». PloS One 15, no 9 (2020): e0232566. https://doi.org/10.1371/journal.pone.0232566.
González-Fuente, Manuel, Sébastien Carrère, Dario Monachello, Benjamin G. Marsella, Anne-Claire Cazalé, Claudine Zischek, Raka M. Mitra, et al. « EffectorK, a Comprehensive Resource to Mine for Ralstonia, Xanthomonas, and Other Published Effector Interactors in the Arabidopsis Proteome ». Molecular Plant Pathology 21, no 10 (octobre 2020): 1257‑70. https://doi.org/10.1111/mpp.12965.
Gluck-Thaler, Emile, Aude Cerutti, Alvaro L. Perez-Quintero, Jules Butchacas, Verónica Roman-Reyna, Vishnu Narayanan Madhavan, Deepak Shantharaj, et al. « Repeated Gain and Loss of a Single Gene Modulates the Evolution of Vascular Plant Pathogen Lifestyles ». Science Advances 6, no 46 (novembre 2020): eabc4516. https://doi.org/10.1126/sciadv.abc4516.
Arroyo-Velez, Noe, Manuel González-Fuente, Nemo Peeters, Emmanuelle Lauber, et Laurent D. Noël. « From Effectors to Effectomes: Are Functional Studies of Individual Effectors Enough to Decipher Plant Pathogen Infectious Strategies? » PLoS Pathogens 16, no 12 (décembre 2020): e1009059. https://doi.org/10.1371/journal.ppat.1009059.
2019
Cerutti, Aude, Alain Jauneau, Patrick Laufs, Nathalie Leonhardt, Martin H. Schattat, Richard Berthomé, Jean-Marc Routaboul, et Laurent D. Noël. « Mangroves in the Leaves: Anatomy, Physiology, and Immunity of Epithemal Hydathodes ». Annual Review of Phytopathology 57 (25 août 2019): 91‑116. https://doi.org/10.1146/annurev-phyto-082718-100228.
2018
Denancé, Nicolas, Boris Szurek, Erin L. Doyle, Emmanuelle Lauber, Lisa Fontaine-Bodin, Sébastien Carrère, Endrick Guy, et al. « Two Ancestral Genes Shaped the Xanthomonas Campestris TAL Effector Gene Repertoire ». The New Phytologist 219, no 1 (juillet 2018): 391‑407. https://doi.org/10.1111/nph.15148.
2017
Cerutti, Aude, Marie-Christine Auriac, Laurent D. Noël, et Alain Jauneau. « Histochemical Preparations to Depict the Structure of Cauliflower Leaf Hydathodes ». Bio-Protocol 7, no 20 (20 octobre 2017): e2452. https://doi.org/10.21769/BioProtoc.2452.
Cerutti, Aude, et Alain Jauneau. « Capturing Z-Stacked Confocal Images of Living Bacteria Entering Hydathode Pores of Cauliflower ». Bio-Protocol 7, no 20 (20 octobre 2017): e2451. https://doi.org/10.21769/BioProtoc.2451.
Cerutti, Aude, Alain Jauneau, Marie-Christine Auriac, Emmanuelle Lauber, Yves Martinez, Serge Chiarenza, Nathalie Leonhardt, Richard Berthomé, et Laurent D. Noël. « Immunity at Cauliflower Hydathodes Controls Systemic Infection by Xanthomonas Campestris Pv Campestris ». Plant Physiology 174, no 2 (juin 2017): 700‑716. https://doi.org/10.1104/pp.16.01852.
2016
Boulanger, Alice, et Laurent D. Noël. « Xanthomonas Whole Genome Sequencing: Phylogenetics, Host Specificity and Beyond ». Frontiers in Microbiology 7 (2016): 1100. https://doi.org/10.3389/fmicb.2016.01100.
Denancé, Nicolas, Thomas Lahaye, et Laurent D. Noël. « Editorial: Genomics and Effectomics of the Crop Killer Xanthomonas ». Frontiers in Plant Science 7 (2016): 71. https://doi.org/10.3389/fpls.2016.00071.
Jacques, Marie-Agnès, Matthieu Arlat, Alice Boulanger, Tristan Boureau, Sébastien Carrère, Sophie Cesbron, Nicolas W. G. Chen, et al. « Using Ecology, Physiology, and Genomics to Understand Host Specificity in Xanthomonas ». Annual Review of Phytopathology 54 (4 août 2016): 163‑87. https://doi.org/10.1146/annurev-phyto-080615-100147.
Robène, I., S. Bolot, O. Pruvost, M. Arlat, L. D. Noël, S. Carrère, M.-A. Jacques, R. Koebnik, et L. Gagnevin. « High-Quality Draft Genome Sequences of Two Xanthomonas Pathotype Strains Infecting Aroid Plants ». Genome Announcements 4, no 5 (1 septembre 2016): e00902-16. https://doi.org/10.1128/genomeA.00902-16.
2015
Bolot, Stéphanie, Aude Cerutti, Sébastien Carrère, Matthieu Arlat, Marion Fischer-Le Saux, Perrine Portier, Stéphane Poussier, Marie-Agnes Jacques, et Laurent D. Noël. « Genome Sequences of the Race 1 and Race 4 Xanthomonas Campestris Pv. Campestris Strains CFBP 1869 and CFBP 5817 ». Genome Announcements 3, no 5 (17 septembre 2015): e01023-15. https://doi.org/10.1128/genomeA.01023-15.
Pesce, Céline, Stéphanie Bolot, Edwige Berthelot, Claude Bragard, Sébastien Cunnac, Marion Fischer-Le Saux, Perrine Portier, et al. « Draft Genome Sequence of Xanthomonas Translucens Pv. Graminis Pathotype Strain CFBP 2053 ». Genome Announcements 3, no 5 (8 octobre 2015): e01174-15. https://doi.org/10.1128/genomeA.01174-15.
Pesce, Céline, Stéphanie Bolot, Sébastien Cunnac, Perrine Portier, Marion Fischer-Le Saux, Marie-Agnès Jacques, Lionel Gagnevin, et al. « High-Quality Draft Genome Sequence of the Xanthomonas Translucens Pv. Cerealis Pathotype Strain CFBP 2541 ». Genome Announcements 3, no 1 (12 février 2015): e01574-14. https://doi.org/10.1128/genomeA.01574-14.
Roux, Brice, Stéphanie Bolot, Endrick Guy, Nicolas Denancé, Martine Lautier, Marie-Françoise Jardinaud, Marion Fischer-Le Saux, et al. « Genomics and Transcriptomics of Xanthomonas Campestris Species Challenge the Concept of Core Type III Effectome ». BMC Genomics 16 (18 novembre 2015): 975. https://doi.org/10.1186/s12864-015-2190-0.
Wang, Guoxun, Brice Roux, Feng Feng, Endrick Guy, Lin Li, Nannan Li, Xiaojuan Zhang, et al. « The Decoy Substrate of a Pathogen Effector and a Pseudokinase Specify Pathogen-Induced Modified-Self Recognition and Immunity in Plants ». Cell Host & Microbe 18, no 3 (9 septembre 2015): 285‑95. https://doi.org/10.1016/j.chom.2015.08.004.












